Введение
На сегодняшний день задача открытия новых лекарственных препаратов является весьма востребованной и важной для мирового сообщества. Многие заболевания, в том числе с неизвестной этиологией, которые в недавнем прошлом считались неизлечимыми, были изучены с помощью исследований in silico. Компьютерное моделирование лекарственных препаратов в настоящее время является важным инструментом, который может значительно сократить время и затраты, необходимые для разработки новых терапевтических средств. С помощью исследований in silico была значительно ускорена разработка препаратов для терапии многих заболеваний, таких как вирус иммунодефицита человека, хронический миелоидный лейкоз, COVID-19, сахарный диабет, лекарственно-устойчивый туберкулез и др.
Применение алгоритмов глубокого обучения может ускорить и удешевить процесс разработки лекарств. Кроме того, использование методов машинного обучения позволяет получать соединения, отсутствующие в существующих химических базах данных. Таким образом, методы машинного обучения могут помочь разработать новые соединения, которые будут более эффективными ингибиторами данной молекулярной мишени, чем известные в настоящее время лекарственные препараты.
Целью настоящей работы был поиск новых мощных ингибиторов сайта связывания CD4 белка оболочки ВИЧ-1 gp120 с использованием генеративной модели LSTM в сочетании с методами виртуального скрининга на основе фармакофоров и молекулярного моделирования.
Чтобы достичь поставленной цели, были решены следующие задачи: (i) выбор подходящей модели генеративной нейронной сети, основанной на удобстве входных данных и скорости обучения и генерации; (ii) формирование обучающего набора данных, включающего соединения, способные специфически и эффективно взаимодействовать с CD4-связывающим сайт gp120; подготовка терапевтической мишени и молекулярный докинг; (iii) обучение модели автоэнкодера; (iv) генерация ряда новых низкомолекулярных соединений, потенциально активных по отношению к gp120; (v) оценка значений энергии связывания полученных соединений с gp120 путем молекулярного докинга; (vi) анализ данных молекулярного докинга и отбор ведущих соединений, перспективных для разработки новых эффективных ингибиторов белка оболочки gp120 ВИЧ-1.
Архитектура автоэнкодера LSTM
В выбранной модели автоэнкодера слои LSTM используются для обработки входных данных, после чего состояния из этих слоев объединяются на объединяющем слое, образуя вложения. Эти вложения предварительно обрабатываются полностью подключенным слоем и объединяются на объединяющем слое с данными о нейроне, ответственном за энергию связи соединения, образуя скрытое пространство.
Полученные элементы скрытого пространства передаются в декодер, где они используются для создания состояний уровня LSTM. Для этого данные из скрытого пространства передаются на два независимых полностью подключенных уровня, а затем их выходные данные передаются в виде краткосрочных и долгосрочных состояний на уровень LSTM. Входные данные уровня LSTM совпадают с теми, которые ожидаются на выходе нейронной сети. Это позволяет повысить производительность декодера и ускорить обучение модели. Выходные данные с уровня LSTM далее поступают на полностью подключенный уровень с функцией активации softmax, которая используется для получения вероятностей следующего символа в выходных данных. Для всех остальных полностью подключенных слоев использовалась функция активации ReLU, а для слоев LSTM – функция активации tanh. Общая схема этой модели автоэнкодера на основе LSTM показана на рис. 1.
Подготовка входных данных
Перед обучением нейронной сети был сформирован обучающий набор данных из 77 184 низкомолекулярных соединений. С этой целью кристаллическая структура мощного ингибитора проникновения ВИЧ-1 NBD-14204, связанного с белком вирусной оболочки gp120 (PDB ID: 8F9Z), была использована для создания фармакофорной модели этого противовирусного средства, проведения виртуального скрининга химических баз данных на основе фармакофоров (Pharmit; https://pharmit.csb.pitt.edu) и молекулярного докинга (AutoDock Vina; https://vina.scripps.edu ).
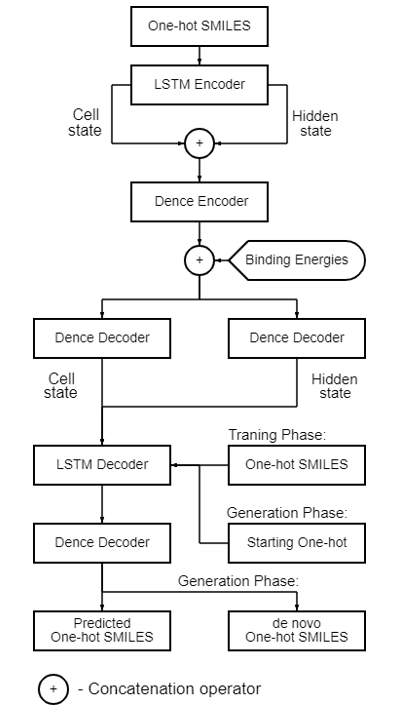
Рис. 1. Архитектура модели автоэнкодера на основе LSTM
Модель фармакофора представляет собой набор стерических и электронных свойств, необходимых для обеспечения оптимального молекулярного взаимодействия с конкретной биологической мишенью и запуска (или блокирования) ее биологического ответа.
Для запуска виртуального скрининга файл, описывающий комплекс NBD-14204/gp120 в crystal (PDB ID: 8F9Z), был загружен из базы данных PDB и обработан с использованием программного пакета UCSF Chimera и веб-сервера SWISS-MODEL – инструмента открытого доступа для автоматизированного сравнительное моделирование трехмерных белковых структур. Виртуальный скрининг проводился с использованием химических баз данных веб-сервиса Pharmit, в частности CHEMBL30, ChemDiv, ChemSpace, MCULE-ULTIMATE, MolPort, NCI, LabNetwork и ZINC-15. В то же время было использовано «правило пяти» Липински с последующей фильтрацией соединений по среднеквадратичным отклонениям между признаками запроса и признаками попадающего соединения и ограничением значения энергии связи.
Перед молекулярным докингом атомы водорода были добавлены к восстановленной структуре ВИЧ-1 gp120 с помощью программного пакета USCF Chimera. Неполярные атомы водорода были удалены из структуры белка с помощью AutoDockTools-1.5.7, а исходный файл был переведен из формата .pdb в формат .pdbqt, необходимый для AutoDock Vina. Молекулярный докинг, автоматизированный компьютерный алгоритм, позволяющий определить положение лиганда в активном центре белка и рассчитать значения свободной энергии связывания, был выполнен в приближении жесткого рецептора и гибких лигандов. Ячейка сетки для стыковки включала сайт связывания CD4 gp120 и имела следующие параметры: ΔX = 21,5 Å, ΔY = 21 Å, ΔZ = 25 Å с центром в X = 2,4 Å, Y = 16,57 Å, Z = 11,95 Å. Значение параметра исчерпываемости, который определяет количество прогонов отдельных образцов, было равно 100.
После молекулярного докинга данные были перенесены в формат SMILES canon и для каждой строки было сгенерировано пять строк в формате SMILES, где каждая строка SMILES начиналась с атома, отличающегося от других в исходной структуре, что позволило расширить набор обучающих данных до 385 920 соединений. Кроме того, соединения, в которых символы встречались реже, чем в 0,07 % случаев, были удалены из обучающего набора данных.
Чтобы предоставить входные данные, строки SMILES были преобразованы методом однократного кодирования в матрицы, в которых первым символом добавляется «!», а символы «E» добавляются после фактической последовательности SMILES в конец строки. В этих матрицах индексы в строке из 120 символов расположены горизонтально, а словарь символов, составленный на основе обучающего набора данных, расположен вертикально.
Обучение автоэнкодера
Категориальная перекрестная энтропия (CCE) использовалась в качестве функции потерь и рассчитывалась по формуле
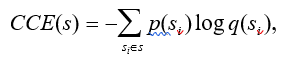
где p(sRiR) и q(sRiR) – истинная и прогнозируемая вероятности генерации сим- вола sRi Rстрок соответственно.
Данная функция является классическим подходом, используемым в машинном обучении для решения задач многоклассовой классификации. Для ее оптимальной работы требуется выбрать веса таким образом, чтобы эта функция достигала минимального значения на входных данных обучения.
В качестве оптимизатора использовался метод ADAM, классический алгоритм адаптивного градиентного спуска с использованием моментов, со значением интенсивности обучения ε = 0,008.
После завершения процесса обучения была проведена оценка модели. На рис. 2 показаны графики функции потерь для 100 периодов обучения.
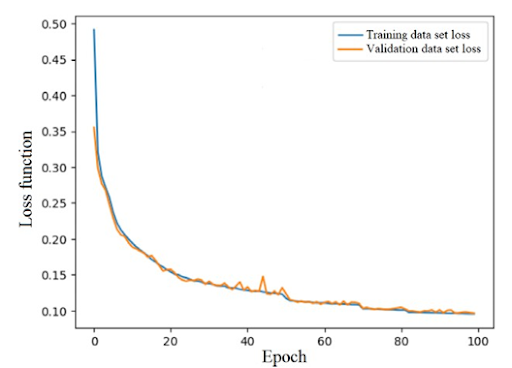
Рис. 2. Потери при обучении и валидации для модели автоэнкодера на основе LSTM
Генерация соединений
Чтобы сгенерировать новые данные после разработки полноценного автоэнкодера, процесс был разделен на три модели. Первая модель выполняет функцию преобразования исходных данных в вектор скрытого пространства, который является кодирующей частью автоэнкодера. Используя эту модель, можно получить представление о скрытом пространстве из набора проверочных выборок, передав его на вход модели. Вторая модель преобразует данные из скрытого пространства в векторы состояния для слоя LSTM третьей модели, проходя через полностью подключенный слой. Третья модель состоит из входного и LSTM-слоев, а также из полностью подключенного слоя, имеющего веса с той же размерностью, что и автоэнкодер, которые инициализируются путем передачи весов из обученной нейронной сети. Модель принимает в качестве входных данных один однократно закодированный символ и вектор начальных состояний для слоя LSTM из предыдущей модели и выводит один закодированный символ, который предположительно является следующим в сгенерированной строке. Генерация начинается с начала строкового символа «!» и заканчивается, как только встречается конец строкового символа «E».
Результаты
С помощью автоэнкодера на основе LSTM было сгенерировано 46 846 новых соединений в формате SMILES. Эти SMILES были очищены от дубликатов, проверены на достоверность и интерпретируемость с помощью модуля RDKit (http://www.rdkit.org/) и преобразованы в двумерные и трехмерные химические структуры.
Значения свободной энергии связывания с белком gp120 ВИЧ-1 были оценены для 5000 молекул, выбранных случайным образом из выборки полученных соединений. Результаты оценки энергии связи для исходной и сгенерированной молекул представлены на рис. 3 в виде двух гистограмм.
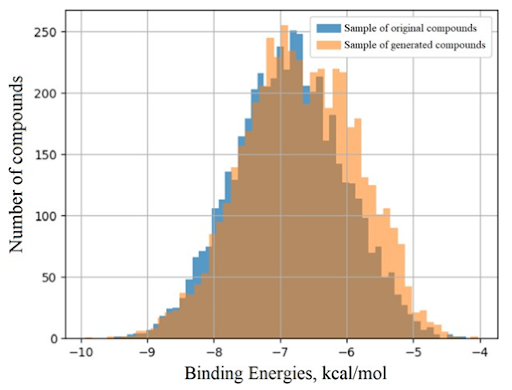
Рис. 3. Гистограммы распределения количества соединений по энергии связи для образца исходного и полученного соединений
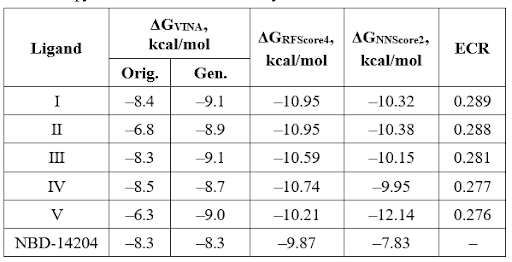
Согласно данным молекулярного докинга, для 159 из 5000 полученных соединений значения свободной энергии связывания с gp120 были близки или ниже, чем у эталонного ингибитора ВИЧ-1 NBD-14204 (для NBD-14204 это значение было равно 8,3 ккал/моль). Кроме того, была проведена более точная оценка аффинности связывания белок-лиганд для 5000 выбранных соединений с использованием функций оценки NNScore 2.0 и RF-Score-4.
Результаты, полученные для пяти лучших по оценке ECR соединений и NBD-14204, приведены в таблице.
Значения функций оценки для пяти лучших соединений и NBD-14204
Растворимость в воде, синтетическая доступность и токсичность были оценены для 100 полученных соединений с наилучшими значениями ECR с использованием платформы SwissADME, бесплатного веб-инструмента для оценки лекарственных свойств химических соединений (http://swissadme.ch). Согласно прогнозам SwissADME, 84 соединения удовлетворяют физико-химическим параметрам, обычно используемым в качестве основных фильтров для отбора молекул на предмет их способности быть эффективными лекарственными средствами.
Химические структуры трех соединений, занявших первое место, и NBD-14204 показаны на рис. 4.
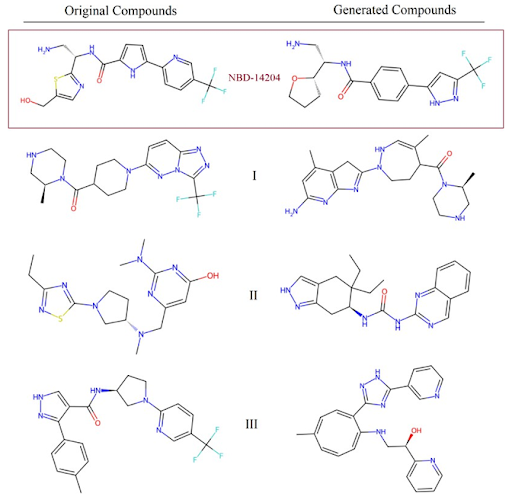
Рис. 4. Пример полученных соединений
Заключение
Генеративная нейронная сеть на основе LSTM была перепрофилирована для разработки потенциальных ингибиторов гликопротеина оболочки ВИЧ-1 gp120, играющего ключевую роль в прикреплении вируса к клеточному рецептору CD4. Эта генеративная модель автоэнкодера была обучена и протестирована, а также проанализированы результаты ее рабо- ты. Во время валидации нейронной сети было сгенерировано 46 846 молекул, и их ингибирующий потенциал был оценен с помощью инструментов молекулярного докинга.
В результате были идентифицированы 84 соединения, представляющие большой интерес для дальнейших исследований, которые будут использоваться в качестве базовых структур для разработки новых мощных противовирусных средств, способных остановить распространение ВИЧ/СПИДа.